Создание контрольного списка в приложении Word
Word для Microsoft 365 Word для Интернета Word 2021 Word 2019 Word 2016 Word 2013 Word 2010 Word 2007 Еще…Меньше
Отображение вкладки «Разработчик»
Если вкладка «Разработчик» не отображается, см. вкладку «Разработчик».
Составление списка
-
Введите список.
-
Перейдите в «Разработчик» и выберите «Контрольный поле» в начале первой строки.
-
Скопируйте и вкопируйте поле с квадратиконом в начале каждой строки.
-
Чтобы поместить в поле X (или другой символ) снимок, выберите его.
О том, как изменить X по умолчанию, см. в подменю «Изменение почтовых ящиков».
Добавление отступа
Если некоторые элементы списка имеют длину более одной строки, для их выравнивания используйте отступы.
- org/ListItem»>
Выделите список. Маркеры или номера при этом не выглядят выделенными.
-
На ленте «Главная»выберите в диалоговом окне «Абзац» .
-
В разделе Отступ в поле Слева введите значение 0,25.
-
-
В поле На по умолчанию указывается значение 0,5 дюйма.
 Если вы хотите, чтобы у дополнительных строк был другой отступ, измените его.
Если вы хотите, чтобы у дополнительных строк был другой отступ, измените его. -
По завершению выберите «ОК».
Блокировка текста и элементов управления для предотвращения изменений
-
Выберите список и их почтовые ящики.
-
Перейдите в группу > для >разработчиков.
Чтобы разблокировать список, выберем его и выберите «Группировать>разгруппировать.

Создание списка только для печати
-
Перейдите в «Главная» и выберите стрелку вниз рядом с кнопкой «Маркеры».
-
В меню выберите пункт «Определить новый маркер».
-
Выберите «Символ» и найдите символ квадратика. Если вы изначально его не видите, измените шрифт на Wingdings или Segoe UI Symbol.

-
Дважды выберите «ОК» и создайте список.
Создание контрольного списка, который можно отмечать в Word
Чтобы создать список, который можно поохить в Word, добавьте в документ элементы управления содержимым для контрольных элементов.Отображение вкладки «Разработчик»
Дополнительные сведения о том, не отображается ли вкладка «Разработчик» в Word, см. в этом случае.
Составление списка
- org/ItemList»>
-
Создайте список.
Поместите курсор в начало первой строки.
-
Перейдите на вкладку «Разработчик» и нажмите кнопку «Квадрат».
-
Если после этого квадрата нужно вжать пробел, нажмите tab или ПРОБЕЛ.
Используйте знак табуляции, если вы хотите отформатировали элемент списка с помощью отступа, как Word для маркеров.

-
Выберите и скопируйте (⌘+C) квадратику и все вкладки или пробелы.
-
Поместите курсор в начало каждого элемента списка и вдайте (⌘+V) скопированные квадраты и символы.
-
В каждой строке списка в каждой строке в этой строке будет в конце списка.
Добавление отступа
Если некоторые элементы списка длиннее одной строки, для их выравнивания используйте отступы.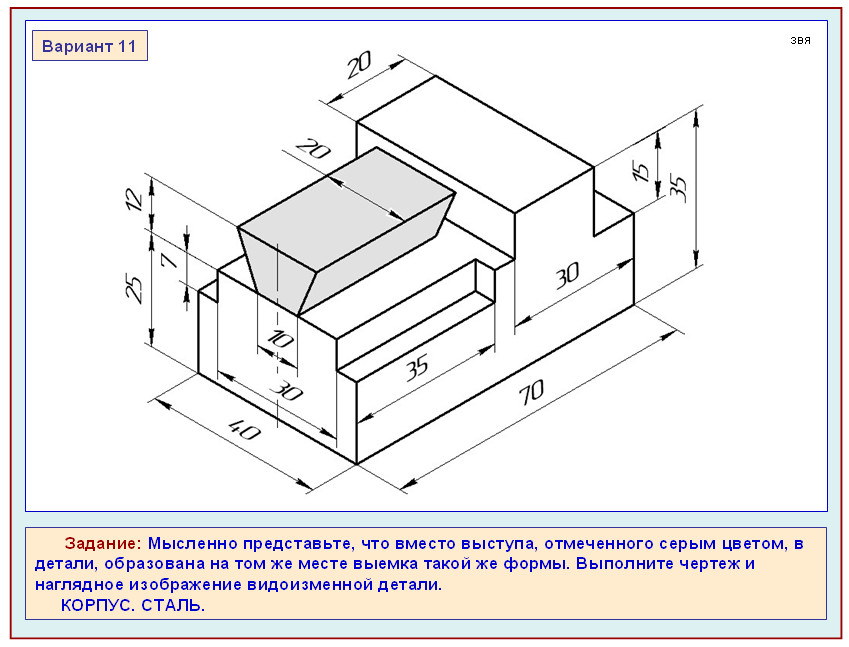
-
Выделите список.
-
Перейдите к пункту «Формат» и выберите «Абзац».
-
В разделе Отступ в поле Слева введите значение 0,25.
-
В списке «Специальный» выберите «Висячий».
В поле На по умолчанию указывается значение 0,5 дюйма.
 Если вы хотите, чтобы у дополнительных строк был другой отступ, измените его.
Если вы хотите, чтобы у дополнительных строк был другой отступ, измените его.
Печать только контрольного списка
Маркеры или номера в списке можно заменить другим символом, например, если в печатной версии документа может быть проверено поле.
-
Выделите весь список.
На ленте выберите «Главная» и стрелку рядом с маркерами.
- org/ListItem»>
Выберите «Определить новый маркер».
-
Выберите «Маркер»и выберите символ из таблицы символов.
Чтобы увидеть другие варианты, в поле Шрифт выберите символьный шрифт, например Wingdings.
-
Выберите символ, который нужно использовать, например открытый ) или объемный ().
-
Выберите «ОК»,а затем еще раз выберите «ОК».
Word в Интернете поддерживает только бумажную форму контрольного списка.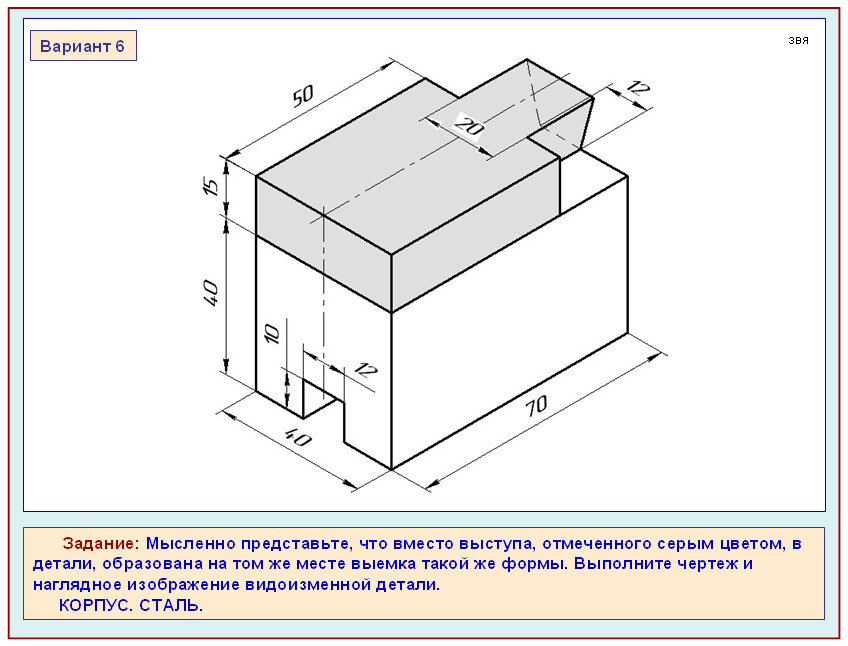
-
Напишите список и выберите его.
-
На вкладке «Главная» выберите стрелку вниз рядом со списком «Маркеры», чтобы открыть библиотеку маркеров, а затем выберите символ контрольного списка.
MS Word Списки
Правила оформления различных документов иногда требуют наличия в документах списков. Принято выделять три типа списков:
Маркированные списки (или списки-бюллетени) используются при перечислении или выделении отдельных фрагментов текста. В качестве примера можно использоваться данный фрагмент текста, оформленный с помощью средств маркированного списка.
Нумерованные списки полезны в тех случаях, когда нужно определить порядок изложения.
Многоуровневые (или иерархические) списки, т.е. имеющие несколько уровней. В таких списках допустимы как нумерованные элементы, так и символы маркера.
Создание маркированных и нумерованных списков
Маркированный (нумерованный) список можно создать с помощью окна диалога Список и панели Форматирование. Для создания маркированного списка с помощью окна диалога Список необходимо выполнить следующие действия:
1. Выделить несколько абзацев текста, предназначенных для создания списка.
2. Выбрать команду Формат – Список. Откроется окно диалога Список.
3. Перейти на вкладку Маркированный (Нумерованный) (рис. 4.24).
4. Выбрать список из представленных образцов. Нажать ОК.
Нажать ОК.
Для продолжения списка необходимо поместить курсор в конце последнего абзаца списка и нажать клавишу Enter.
Для создания списков на панели инструментов Форматирование предназначены две кнопки:
Маркированный список
Создает маркированный список
Нумерованный список
Создает нумерованный список
Рис. 4.24. Окно диалога Список. Вкладка Маркированный.
При создании списков с помощью кнопок панели инструментов Форматирование параметры их будут заданы по умолчанию.
Для завершения списка необходимо установить курсор в пределах абзаца, начиная с которого нужно отменить список, и нажать кнопку, соответствующую списку (нумерованный или маркированный).
Изменение формата маркированного списка
С помощью окна диалога Список можно изменить следующие параметры маркированного списка:
Символ маркера на любой символ из шрифтов, установленных на компьютере.
Величину выступа.
Расстояние между символом маркера и текстом.
Выравнивание.
Для этих целей необходимо войти в меню Формат – Список и нажать кнопку Изменить, после чего откроется окно диалога Изменить маркированный список, представленное на рис. 4.25.
Рис. 4.25. Окно диалога Изменить маркированный список
Ниже приведен состав его опций:
| Опции | Назначение |
Символ маркера |
|
| Шрифт | Предлагает варианты различных шрифтов для выбранного символа |
| Маркер | Позволяет осуществить выбор варианта символа маркера |
Положение маркера |
|
| Отступ | Определяет выравнивание и положение маркера относительно левой границы текста |
Положение от текста |
|
| Отступ | Устанавливает величину отступа от правого края маркера до начала текста |
| Образец | Позволяет визуально оценить, как будет выглядеть список после внесения в него изменений |
Изменение формата нумерованного списка
Нумерованные списки создаются на вкладке Нумерованные (рис. 4.24). Если предложенные форматы нумерованного списка не подходят для оформления конкретного документа, можно создать собственный формат списка, нажав кнопку Изменить в диалоговом окне Список на вкладке Нумерованный. При этом откроется окно диалога Изменение нумерованного списка (рис. 4.26).
4.24). Если предложенные форматы нумерованного списка не подходят для оформления конкретного документа, можно создать собственный формат списка, нажав кнопку Изменить в диалоговом окне Список на вкладке Нумерованный. При этом откроется окно диалога Изменение нумерованного списка (рис. 4.26).
Рис. 4.26. Окно диалога Изменить нумерованный список
Создание многоуровневых списков
Во многих видах документов встречаются более сложные типы списков. Это так называемые многоуровневые списки. В них также используется либо маркировка, либо нумерация, а иногда и сочетание того и другого. Но главное их отличие от предыдущих типов списков – наличие нескольких уровней. В зависимости от уровня для каждого абзаца устанавливается символ или номер.
Рис. 4.27. Вкладка Многоуровневые окна диалога Список.
Многоуровневые списки отличаются от обычных наличием отступов. Определенный отступ принимается за уровень такого списка.
Определенный отступ принимается за уровень такого списка.
Создание многоуровневого списка возможно только с помощью окна диалога Список. Причем уровни задает сам исполнитель, устанавливая определенные отступы. Word анализирует отступы и, в зависимости от их величины, нумерует, создавая уровни.
Можно создавать измененные форматы нумерации для различных уровней многоуровневого списка, однако в одном документе допустим только один формат многоуровневого списка.
Для создания многоуровневого списка необходимо выполнить следующие действия:
1. Определить фрагмент документа, который предполагается оформить в виде списка.
2. Создать отступы каждого абзаца, воспользовавшись кнопкой Увеличить отступ на панели инструментов Форматирование (при создании отступов нельзя использовать табуляцию).
3. Выделить выбранный фрагмент текста.
4. Выбрать команду Формат – Список. В окне диалога Список перейти на вкладку Многоуровневый (рис. 4.27).
5. Выбрать нужный тип многоуровневого списка из предложенных вариантов. Нажать ОК.
Работа в Microsoft Word XP
Главная / Офисные технологии / Работа в Microsoft Word XP / Тест 4
Упражнение 1:
Номер 1
С помощью какого элемента можно создать маркированный список?
Ответ:
 (1) 1 
 (2) 2 
 (3) 3 
 (4) 4 
Номер 2
С помощью какого элемента можно создать нумерованный список?
Ответ:
 (1) 1 
 (2) 2 
 (3) 3 
 (4) 4 
Номер 3
С помощью какого элемента можно создать многоуровневый список?
Ответ:
 (1) 1 
 (2) 2 
 (3) 3 
 (4) 4 
Номер 4
Что можно нумеровать с использованием нумерованного списка?
Ответ:
 (1) слова в абзаце 
 (2) предложения в абзаце 
 (3) строки в абзаце 
 (4) абзацы в тексте 
Упражнение 2:
Номер 1
Что можно маркировать с использованием маркированного списка?
Ответ:
 (1) слова в абзаце 
 (2) предложения в абзаце 
 (3) строки в абзаце 
 (4) абзацы в тексте 
Номер 2
Можно ли создать непрерывный нумерованный список на нескольких страницах?
Ответ:
 (1) нет 
 (2) да, но только если весь текст оформлен шрифтом одного размера 
 (3) да, но только если текст не имеет разделов 
 (4) да 
Номер 3
Можно ли создать непрерывный маркированный список на нескольких страницах?
Ответ:
 (1) нет 
 (2) да, но только если весь текст оформлен шрифтом одного размера 
 (3) да, но только если текст не имеет разделов 
 (4) да 
Номер 4
С помощью какого элемента можно понизить уровень выделенного текста для создания многоуровневого списка?
Ответ:
 (1) 1 
 (2) 2 
 (3) 3 
 (4) 4 
Упражнение 3:
Номер 1
С помощью какого элемента можно повысить уровень выделенного текста для исправления многоуровневого списка?
Ответ:
 (1) 1 
 (2) 2 
 (3) 3 
 (4) 4 
Номер 2
Что устанавливает выбранный список?
Ответ:
 (1) маркировку слов в выделенных абзацах выбранным символом 
 (2) маркировку выделенных абзацев выбранным символом 
 (3) маркировку предложений в выделенных абзацах выбранным символом 
 (4) размещение выбранных символов в правом верхнем углу каждой страницы 
Номер 3
Что устанавливает выбранный список?
Ответ:
 (1) нумерацию слов в выделенных абзацах 
 (2) нумерацию выделенных абзацев 
 (3) нумерацию предложений в выделенных абзацах 
 (4) нумерацию страниц 
Номер 4
Как можно сортировать список?
Ответ:
 (1) с помощью команды Таблица/Сортировка 
 (2) с помощью команды Формат/Регистр 
 (3) с помощью команды Сервис/Настройка 
 (4) с помощью команды Сервис/Параметры 
Упражнение 4:
Номер 1
С помощью какого элемента можно удалить нумерацию многоуровневого списка?
Ответ:
 (1) 1 
 (2) 2 
 (3) 3 
 (4) 4 
Номер 2
Может ли шрифт нумерации отличаться от шрифта нумеруемых абзацев?
Ответ:
 (1) нет 
 (2) да, но только начертанием 
 (3) да, но только размером 
 (4) да 
Номер 3
Для чего можно использовать кнопку Сброс?
Ответ:
 (1) для удаления нумерации списка 
 (2) для преобразования в маркированный список 
 (3) для преобразования в многоуровневый список 
 (4) для восстановления параметров списка, принятых по умолчанию 
Номер 4
Для чего можно использовать кнопку Изменить?
Ответ:
 (1) для изменения начального номера списка 
 (2) для изменения параметров шрифта номеров списка 
 (3) для изменения параметров шрифта абзацев списка 
 (4) для изменения отступа текста от левого поля страницы 
Упражнение 5:
Номер 1
Для чего можно использовать кнопку Сброс?
Ответ:
 (1) для удаления маркеров 
 (2) для преобразования в нумерованный список 
 (3) для преобразования в многоуровневый список 
 (4) для восстановления маркеров, принятых по умолчанию 
Номер 2
Для чего можно использовать кнопку Изменить?
Ответ:
 (1) для изменения цвета маркера 
 (2) для выбора другого маркера 
 (3) для изменения отступа текста от правого поля страницы 
 (4) для изменения отступа текста от левого поля страницы 
Номер 3
С помощью какого элемента можно удалить маркеры?
Ответ:
 (1) 1 
 (2) 2 
 (3) 3 
 (4) 4 
Номер 4
С помощью какого элемента можно удалить нумерацию?
Ответ:
 (1) 1 
 (2) 2 
 (3) 3 
 (4) 4 
Упражнение 6:
Номер 1
Какие элементы линейки можно использовать для задания абзацного отступа (красной строки)?
Ответ:
 (1) 1 
 (2) 2 
 (3) 3 
 (4) 4 
Номер 2
Какой элемент диалогового окна используется для задания абзацного отступа (красной строки)?
Ответ:
 (1) 1 
 (2) 2 
 (3) 3 
 (4) 4 
Номер 3
Какой межстрочный интервал не зависит от размера шрифта?
Ответ:
 (1) 1 (Одинарный) 
 (2) 2 (Минимум) 
 (3) 3 (Точно) 
 (4) 4 (Множитель) 
Номер 4
В каких единицах установлен интервал в раскрывающемся списке междустрочный?
Ответ:
 (1) в сантиметрах 
 (2) в дюймах 
 (3) в пунктах 
 (4) в строках 
Упражнение 7:
Номер 1
Что устанавливает Выступ 1,25 см?
Ответ:
 (1) смещение первой строки абзаца на 1,25 см вправо от левого поля 
 (2) смещение первой строки абзаца на 1,25 см влево от левого поля 
 (3) смещение всего абзаца кроме первой строки абзаца на 1,25 см вправо от левого поля 
 (4) смещение всего абзаца кроме первой строки абзаца на 1,25 см влево от левого поля 
Номер 2
Что устанавливает Отступ слева 2 см?
Ответ:
 (1) смещение всего абзаца на 2 см влево от левого поля 
 (2) смещение всего абзаца на 2 см вправо от левого поля 
 (3) размер левого поля страницы 
 (4) смещение только первой строки абзаца на 2 см влево от левого поля 
Номер 3
Что устанавливает Интервал перед 6 пт?
Ответ:
 (1) смещение первой строки абзаца на 6 пт вправо от левого поля 
 (2) увеличение межстрочного интервала на 6 пт 
 (3) смещение абзаца на 6 пт см влево от левого поля 
 (4) увеличение межстрочного интервала перед абзацем на 6 пт 
Номер 4
Какие элементы можно использовать для изменения межстрочного интервала?
Ответ:
 (1) 1 
 (2) 2 
 (3) 3 
 (4) 4 
Упражнение 8:
Номер 1
Какие элементы можно использовать для выравнивания абзаца по ширине страницы?
Ответ:
 (1) 1 
 (2) 2 
 (3) 3 
 (4) 4 
Номер 2
Чем отличается выравнивание абзаца По ширине от выравнивания абзаца По левому краю?
Ответ:
 (1) ничем 
 (2) при выравнивании абзаца По ширине левый край выравнивается по левому полю страницы, а правый — по правому полю 
 (3) при выравнивании абзаца По ширине левый край выравнивается по левому полю страницы, а величина правого поля автоматически подбирается так, что бы абзац не выходил за его пределы 
 (4) при выравнивании абзаца По ширине подбирается такой межстрочный интервал, чтобы абзац не выходил за пределы полей страницы документа 
Номер 3
За счет изменения каких параметров происходит выравнивание абзаца По ширине?
Ответ:
 (1) за счет изменения интервалов между символами текста 
 (2) за счет изменения интервалов между словами 
 (3) за счет изменения размера шрифта 
 (4) за счет изменения межстрочных интервалов 
Номер 4
Для установки границ абзаца применяются элементы панели инструментов Таблицы и границы.В каком порядке следует использовать указанные элементы?
Ответ:
 (1) 1 — 2 — 3 — 4 
 (2) 2 — 1 — 3 — 4 
 (3) 4 — 1 — 3 — 2 
 (4) 1 — 3 — 2 — 4 
Номер 5
Что можно использовать для установки границ абзаца?
Ответ:
 (1) панель инструментов Таблицы и границы 
 (2) панель инструментов Рамки 
 (3) команду Формат/Границы и заливка 
 (4) команду Формат/Рамки 
Упражнение 9:
Номер 1
Какие элементы можно использовать для удаления границ абзаца?
Ответ:
 (1) 1 
 (2) 2 
 (3) 3 
 (4) 4 
Номер 2
Какие элементы можно использовать для установки границ абзаца?
Ответ:
 (1) 1 
 (2) 2 
 (3) 3 
 (4) 4 
Номер 3
Какие элементы можно использовать для установки границ абзаца?
Ответ:
 (1) 1 
 (2) 2 
 (3) 3 
 (4) 4 
 (5) нет правильного ответа 
Номер 4
Какие элементы можно использовать для выбора цвета границ абзаца?
Ответ:
 (1) 1 
 (2) 2 
 (3) 3 
 (4) 4 
Упражнение 10:
Номер 1
Что изменяется при установке цвета заливки?
Ответ:
 (1) цвет текста 
 (2) цвет фона текста 
 (3) цвет всей страницы 
 (4) цвет полей страницы 
Номер 2
Какие команды можно использовать для заливки текста?
Ответ:
 (1) Формат/Границы и заливка 
 (2) Формат/Фон 
 (3) Формат/Автоформат 
 (4) Сервис/Настройка 
Номер 3
Можно ли изменить расстояние от границы до обрамляемого текста?
Ответ:
 (1) да, при обрамлении любого фрагмента 
 (2) да, только при обрамлении абзацев 
 (3) да, только при обрамлении отдельных слов 
 (4) нет 
Номер 4
Какие элементы можно использовать для установки заливки абзаца?
Ответ:
 (1) 1 
 (2) 2 
 (3) 3 
 (4) 4 
Упражнение 11:
Номер 1
Какие элементы можно использовать для изменения расстояния от границы абзаца до текста абзаца?
Ответ:
 (1) 1 
 (2) 2 
 (3) 3 
 (4) 4 
Номер 2
Можно ли для абзаца установить границы с разных сторон разными линиями?
Ответ:
 (1) да 
 (2) да, но линии могут различаться только цветом, а тип должен быть одинаковым 
 (3) да, но линии могут различаться только типом, а цвет должен быть одинаковым 
 (4) нет 
Номер 3
Для какой цели можно использовать кнопку Параметры при обрамлении абзацев?
Ответ:
 (1) для настройки цвета и ширины линии границы 
 (2) для настройки параметров абзаца 
 (3) для изменения расстояния от границы до полей страницы 
 (4) для изменения расстояния от границы до текста абзаца 
Номер 4
Какова максимальная ширина одинарной линии границы?
Ответ:
 (1) 1 пт 
 (2) 3 пт 
 (3) 6 пт 
 (4) 10 пт 
Номер 5
Какова минимальная ширина одинарной линии границы?
Ответ:
 (1) 0 пт 
 (2) 0,1 пт 
 (3) 0,25 пт 
 (4) 0,5 пт 
Главная / Офисные технологии / Работа в Microsoft Word XP / Тест 4
Съемные залитые 5-футовые свесы | NEB
Главная Инструменты и ресурсы Таблицы выбора Разборные залитые 5-футовые свесы
Новые сайты рестрикции могут быть созданы путем лигирования фрагментов ДНК с совместимыми когезионными или тупыми концами. Эти новые сайты рестрикции могут быть созданы:
Эти новые сайты рестрикции могут быть созданы:
- Расщепление с последующим заполнением выступов длиной 5 футов для получения тупых концов.
- Расщепление двумя эндонуклеазами рестрикции, дающими тупые концы.
- Расщепление двумя эндонуклеазами рестрикции, дающими совместимые выступающие части.
В таблице ниже перечислены эндонуклеазы рестрикции, которые генерируют 5′-выступы, которые при заполнении и лигировании приводят к появлению новых сайтов рестрикции. Перечисленные комбинации были идентифицированы с помощью компьютерного анализа, и хотя мы пытались обеспечить их точность, они не обязательно подтверждались экспериментами.
Ферменты рестрикции, которые обладают вырожденной специфичностью распознавания (т.е. распознают более одной последовательности), были исключены из этого списка. Там, где существуют изошизомеры, указан только один член каждого набора. Перечислены только коммерчески доступные ферменты.
Пример:
| Фрагменты EcoRI | Сайты XmnI и AseI | ||
|---|---|---|---|
5′ . ..G ..G | ААТТК…3′ | Заполнить и связать —> | 5′ …ГААТТААТТЦ…3′ |
| 3′ … CTTAA | Г…5′ | 3′ …CTTAATTAAG…5′ |
- Ферменты, выделенные красным цветом, имеют последовательности распознавания из 6 или 8 оснований. Длина последовательности указывается верхним индексом (например, AscI 8 = фреза с 8 основаниями).
- Ферменты в скобках указывают на то, что новая последовательность по-прежнему является субстратом для исходного фермента.
- Верхний индекс 2 указывает на то, что в заполненной/лигированной последовательности были созданы два идентичных сайта. Например, заполнение/лигирование AflII создает последовательность CTTAATTAAG, которая содержит два сайта MseI (TTAA).
| А | Б | Д | Е | Ф | Н | К | М | Н | П | С | Т | Х | Однобуквенный код
| Фермент | Место расщепления | После заполнения/перевязки | Восстановлено |
|---|---|---|---|
| Acc65I | Г/ГТАСС | GGTACGTACC | БсаИ, ХпыЧ5IV, РСАИ, СнаБИ 6 |
| Акил | C/CGC | CCGCGC | (АциИ), БГУИ, ХАИ |
| AcI | АА/ВКТТ | ААКГКГТТ | AflIII, BstUI, MluI 6 |
| АфлII | С/ТТААГ | СТТААТТААГ | МсЭИ 2 , Паци 8 , Цп509И |
| Возраст I * | А/ПГУ | АККГГКЦГГТ | БсиЭИ, (БсрФИ) 2 , ЕАЭИ, ЕАГИ 6 , HaeIII, HpaII |
| АПАЛИ | Г/ТГКАС | ГТГКАТГКАС | Cac8I, NlaIII, NspI, SphI 6 |
| AscI | ГГ/CGCGCC | ГГГКГКГКГККК | (BssHII) 2 , BstUI, Cac8I, HhaI |
| Аври II | C/CTAGG | CCTAGCTAGG | Алюминий, (БФАИ) 2 |
| БамХИ * | Г/ГАТСС | GGATCGATCC | AlwI, ClaI 6 , (DpnII 2 ), TaqI-v2 |
| БКИ * | Т/ГАТКА | TGATCGATCA | гл. 6 , (ДпнИИ 2 ), TaqI-v2 6 , (ДпнИИ 2 ), TaqI-v2 |
| БФАИ | С/ТЕГ | СТАТАГ | SFCI |
| БглИИ | А/ГАТКТ | АГАТЦГАТСТ | Ил 6 , ДпнИИ 2 , TaqI-v2 |
| БСИВИ | C/GTACG | CGTACGTACG | БсаИ, (БсиВИ), ХпыЧ5IV, РСАИ, СнаБИ 6 |
| BspDI/ClaI | АТ/КГАТ | ATCGCGAT | БСтУИ, НИУИ 6 |
| БСПЭИ | Т/ККГГА | TCCGGCCGGA | BsiEI, EaeI, EagI 6 , HaeIII, (HpaII) 2 |
| БСПХИ | Т/КАТГА | TCATGCATGA | (NlaIII) 2 , NsiI 6 |
| БсрГИ | Т/ГТАКА | TGTACGTACA | БСАИ, (РСАИ) 2 , СнаБИ 6 |
| БссХИИ | Г/ЦГКГК | ГКГКГКГКГК | (BssHII), BstUI, Cac8I, HhaI |
| БстБИ | ТТ/CGAA | TTCGCGAA | БСтУИ, НИУИ 6 |
| ДпнИИ/МбоИ/Сау3АИ | /GATC | ГАТЧГАТЦ | ClaI 6 , (ДпнII), TaqI-v2 |
| ЕАГИ * | C/GGCCG | CGGCCGGCCG | BsiEI, BsrFI, Cac8I, EaeI 2 , (EagI 2 ), FseI 8 , HaeIII, HpaII, NaeI |
| EcoRI * | Г/ААТТК | ГАТТААТТС | АсеИ 6 , МсеИ, Цп509И, Хмни 6 |
| ФАТИ | /КАТГ | КАТГКАТГ | БрфБИ 6 , HpyCh5IV, (FatI 2 ) |
| HinP1I | Г/ЦГК | ГКГКГК | BssHII 6 , BstUI, Cac8I, (HhaI) |
| HindIII * | А/АГКТТ | ААГКТАГКТТ | AluI, BfaI, Cac8I, NheI 6 |
| HpaII/MspI | С/CGG | CGCCGG | AciI, BsaJI, BstUI, BtgI, MspA1I, SacII 6 |
| HpyCh5IV | А/ВКТ | АКГКГТ | AflIII, BstUI, MluI 6 |
| Каси | Г/ГГКЦК | ГГГКГКГККК | (БссХИИ 6 ) 2 , (БстУИ) 2 , Cac8I, (HhaI) 2 |
| MfeI * | С/ААТТГ | КААТТААТТГ | АсЭИ 6 , Цп509И 2 |
| МУИ | А/CGCGT | АКГКГКГКГТ | BssHII 6 , BstUI, Cac8I, (HhaI) 2 |
| НарИ | ГГ/КГЦК | ГГГКГККК | AscI 8 , BssHII, BstUI, Cac8I, HhaI |
| NcoI * | С/КАТГГ | ССАТГКАТГГ | NlaIII, NsiI 6 |
| НгоМИВ | Г/ЦКГГК | GCCGGCCGGC | BsiEI, BsrFI, Cac8I, EaeI, EagI 6 , HaeIII, HpaII, (NgoMIV 2 ) |
| NheI * | Г/СТАГК | GCTAGCTAGC | Алюминий, BfaI, Cac8I, (NheI) |
| НотИ * | GC/GGCCGC | GCGGCCGGCCGC | AciI, BsiEI, BsrFI, Cac8I, EaeI, (EagI 6 ) 2 , Фну4ХИ, ФсЭИ 8 , HaeIII, HpaII, NaeI 6 |
| PaeR7I/XhoI | C/TCGAG | CTCGATCGAG | БсиЭИ, ДпниИ, ПвуИ 6 , (TaqI-v2) 2 |
| PCI | А/КАТГТ | АКАТГКАТГТ | HpyCh5V, (NlaIII 2 ), NsiI 6 |
| ПспОМИ | Г/ГГСКК | GGGCCGGCCC | BsrFI, Cac8I, FseI 8 , HaeIII, HpaII, NaeI, Sau96I |
| PspXI | ВК/ТКГАГБ | VCTCGATCGAGB | ПвуИ 6 , (TaqI-v2) 2 |
| Салли * | Г/ТКГАК | GTCGATCGAC | БсиЭИ, ДпниИ, ПвуИ 6 , TaqI-v2 |
Спец. номер * номер * | А/СТАГТ | ACTAGCTAGT | Алюминий, (БФАИ) 2 |
| TaqI-v2 | Т/ЦГА | ТКГКГА | БСтУИ, НИУИ 6 |
| Цп509И | /ААТТ | ААТТААТТ | АсЭИ 6 , МсЭИ, (Цп509И 2 ) |
| XbaI | Т/СТАГА | ТТАГКТАГА | Алюминий, БФАИ |
| XmaI | C/CCGGG | CCCGGCCGGG | БСИЭИ, ЕАЭИ, ЕАГИ 6 , HaeIII, HpaII, NciI, ScrFI |
* Доступны высокоточные версии этих ферментов.
Поиск ферментов рестрикции — NIPPON Genetics EUROPE
Что такое ферменты рестрикции?
Рестрикционные ферменты распознают короткие последовательности ДНК и расщепляют двухцепочечную ДНК в определенном месте узнавания или рядом с ним. Рестрикционные ферменты подразделяются на четыре типа в зависимости от структуры их субъединиц, потребности в кофакторах и специфичности расщепления.
Было обнаружено 3000 различных ферментов рестрикции, которые распознают более 230 различных последовательностей ДНК. Эти ферменты обычно используются для модификации ДНК во всем мире и являются незаменимым инструментом в молекулярном клонировании.
Историческая справка
Основа исследования ферментов рестрикции восходит к работам Лурии и его коллег в начале 1950-х годов [1]. Лурия заметил, что бактериофаг λ может хорошо расти в одном штамме из E.coli (например, E.coli C) , но часто плохо в другом штамме E.coli (например, E.coli K ). Клетка-хозяин ( E.coli K) была известна как рестрикционный хозяин и, по-видимому, обладает способностью снижать биологическую активность фага λ.
Термин «рестриктазы» впервые был упомянут в 1960-х годах в лабораториях Арбера и Мезельсона. Они выяснили, что рестрикция вызвана ферментативным расщеплением ДНК фага. Фермент, участвующий в этом процессе, был назван «ферментом рестрикции» [2, 3]. Ферменты рестрикции, изученные Арбером и Мезельсоном, были ферментами рестрикции типа I, которые расщепляют ДНК в случайных местах вдали от сайта узнавания.
Ферменты рестрикции, изученные Арбером и Мезельсоном, были ферментами рестрикции типа I, которые расщепляют ДНК в случайных местах вдали от сайта узнавания.
В 1970 году Смит и его коллеги выделили и описали первый рестрикционный фермент типа II, Hind II [4]. Ферменты рестрикции II типа гораздо полезнее для лабораторных работ, поскольку они расщепляют ДНК в месте своей узнавающей последовательности. Из-за его важности для молекулярной биологии Смит, Арбер и Натанс разделили Нобелевскую премию 1978 года по медицине и физиологии за открытие рестрикционных ферментов и их применение в молекулярной генетике.
Последовательности распознавания
Все эндонуклеазы рестрикции распознают определенную последовательность ДНК. Последовательность распознавания обычно является палиндромной или частично палиндромной, что означает, что базовая последовательность читается одинаково вперед и назад. Ферменты рестрикции могут расщеплять двухцепочечную ДНК либо в центре обеих цепей с образованием «тупых концов», либо в шахматном порядке, оставляя выступающие части, называемые «липкими концами».
Различные типы рестриктаз
В зависимости от структуры, потребности в кофакторах и специфичности расщепления существует четыре типа рестриктаз (типы I, II, III и IV).
Ферменты рестрикции типа I расщепляют ДНК в произвольном месте, далеком от последовательности распознавания. Для работы этих ферментов требуются как АТФ, так и S-аденозил-L-метионин.
Тип II ферменты рестрикции расщепляют ДНК внутри или рядом с последовательностью распознавания. Эти ферменты не требуют АТФ и не зависят от метилазы. Эндонуклеазы рестрикции наиболее пригодны для ежедневной лабораторной работы. Все наши рестриктазы NIPPON Genetics EUROPE относятся к типу II.
Тип III рестриктазы расщепляют ДНК примерно на 20–25 пар оснований от последовательности распознавания. Для их функционирования требуется как АТФ, так и S-аденозил-L-метионин.
Тип IV рестриктазы расщепляют только модифицированную, обычно метилированную ДНК, в отличие от типов I-III, которые обычно ингибируются метилированием.
Двойное расщепление
Вектор и ДНК-вставка могут быть клонированы путем расщепления двумя разными ферментами рестрикции, что приводит к образованию двух разных концов рестрикции. Эта стратегия предотвращает лигирование вектора без вставки, что приводит к значительному снижению самолигирования и повышению эффективности клонирования. Большинство наших рестрикционных ферментов на 100 % активны в буфере FastCut, что упрощает двойное расщепление. Пожалуйста, посмотрите на Диаграмма двойного дайджеста для проведения двойного дайджеста с четырьмя стандартными буферами.
ДИАГРАММА DOUBLE DIGEST (СКАЧАТЬ)
Activity of Unique FastGene ® Buffer
Nippon Genetics предлагает четыре стандартных буфера, которые максимально поддерживают активность каждого фермента рестрикции в буфере. Однако некоторым эндонуклеазам рестрикции для максимальной активности требуется уникальный буфер. Взгляните на Double Digest Chart для выбора буфера для двойного расщепления, если рестриктазе требуется уникальный буфер. Перечислены активности (%) наиболее часто используемых эндонуклеаз рестрикции в пяти уникальных буферах FastGene ® для EcoRI, BamH l, Acc III, Bal I и Dpn II соответственно. Рестриктазы, если они активны в одном из четырех стандартных буферов, обычно активны в уникальном буфере. Следовательно, можно выполнять двойное переваривание в конкретном уникальном буфере. Если эффективность переваривания низкая из-за неоптимального буфера, увеличьте количество эндонуклеаз рестрикции или инкубируйте в течение более длительного периода времени.
Взгляните на Double Digest Chart для выбора буфера для двойного расщепления, если рестриктазе требуется уникальный буфер. Перечислены активности (%) наиболее часто используемых эндонуклеаз рестрикции в пяти уникальных буферах FastGene ® для EcoRI, BamH l, Acc III, Bal I и Dpn II соответственно. Рестриктазы, если они активны в одном из четырех стандартных буферов, обычно активны в уникальном буфере. Следовательно, можно выполнять двойное переваривание в конкретном уникальном буфере. Если эффективность переваривания низкая из-за неоптимального буфера, увеличьте количество эндонуклеаз рестрикции или инкубируйте в течение более длительного периода времени.
ДВОЙНАЯ ДИАГРАММА (СКАЧАТЬ)
Ссылки
[1] Luria and Human (1952) Ненаследственная, индуцированная хозяином разновидность бактериальных вирусов. J Бактериол ., 557-569.
[2] Арбер и Линн (1969) Модификация и ограничение ДНК. Annu Rev Biochem . , 467-500.
, 467-500.
[3] Meselson and Yuan (1968) Фермент рестрикции ДНК из E. coli. Nature , 1110-1114
[4] Smith and Wilcox (1970) Фермент рестрикции из Hemophilus influenzae. I. Очистка и общие свойства. J Мол Биол ., 379-391.
Разработка ферментов Nt.BtsCI и Nb.BtsCI, вызывающих разрывы, и их применение для получения длинных выступов
Nucleic Acids Res. 2010 март; 38(4): 1294–1303.
Опубликовано в Интернете 2 декабря 2009 г. doi: 10.1093/nar/gkp1092
Информация об авторе Примечания к статье Информация об авторских правах и лицензии Отказ от ответственности и расщепляется ближе к последовательности распознавания. Хотя M.BtsCI демонстрирует 62% идентичность аминокислотной последовательности с M.FokI, эндонуклеазы рестрикции BtsCI и FokI не имеют значительного сходства аминокислотной последовательности. BtsCI принадлежит к группе эндонуклеаз рестрикции IIS, BsmI, Mva1269.I и BsrI, которые несут два разных каталитических сайта в одном полипептиде. Путем инактивации одного из каталитических сайтов путем мутагенеза мы создали варианты BtsCI с надрезами, которые специфически надрезают нижнюю или верхнюю цепь целевого сайта. Последовательно обрабатывая ДНК-мишень соответствующими комбинациями вариантов FokI и BtsCI, мы можем генерировать длинные выступы, подходящие для флуоресцентной маркировки с помощью заполнения концов или других методов, основанных на отжиге комплементарных последовательностей ДНК.
Путем инактивации одного из каталитических сайтов путем мутагенеза мы создали варианты BtsCI с надрезами, которые специфически надрезают нижнюю или верхнюю цепь целевого сайта. Последовательно обрабатывая ДНК-мишень соответствующими комбинациями вариантов FokI и BtsCI, мы можем генерировать длинные выступы, подходящие для флуоресцентной маркировки с помощью заполнения концов или других методов, основанных на отжиге комплементарных последовательностей ДНК.
Большинство эндонуклеаз рестрикции (REases), обычно используемых в молекулярном клонировании, представляют собой ферменты типа IIP, которые имеют хорошо изученную характеристику каталитического мотива PD-X n -E/DXK (где X означает любой аминокислотный остаток). Эти ферменты распознают последовательности палиндромной двухцепочечной ДНК (дцДНК) длиной от 4 до 8 п.н. и расщепляют обе нити в симметричных участках внутри последовательностей распознавания (1,2). Например, димер BamHI связывается с симметричной последовательностью GGATCC. Верхние и нижние разрезы, вероятно, вводятся одновременно после ряда конформационных изменений (изменения интерфейса димера, сужение щели связывания ДНК, развертывание С-концевого плеча и связывание с малой бороздкой, перемещение каталитических остатков более чем на 6 Å). расстояние относительно расщепляемого фосфата (3,4). Кроме того, распознавание и катализ REases типа IIP тесно связаны. Таким образом, было трудно изменить специфичность распознавания при сохранении респектабельной активности (5).Для менее изученных REases типа IIS, распознавание последовательности-мишени и расщепление фосфодиэфирной связи осуществляются отдельными доменами (6–8). Такое разделение функций позволяет распознавать непалиндромы и расщеплять вне сайтов узнавания. Это также приводит к множеству механизмов распознавания ДНК и каталитических механизмов. Например, в AlwI (GGATC 4/5) замена домена расщепления ДНК доменом Nt.BstNBI (GAGTC 4/-) сохраняет специфичность распознавания AlwI, но превращает ее в в эндонуклеазу разрыва верхней цепи (NEase), показывая, что самого домена распознавания ДНК достаточно для специфического связывания с ДНК-мишенью (9).
Верхние и нижние разрезы, вероятно, вводятся одновременно после ряда конформационных изменений (изменения интерфейса димера, сужение щели связывания ДНК, развертывание С-концевого плеча и связывание с малой бороздкой, перемещение каталитических остатков более чем на 6 Å). расстояние относительно расщепляемого фосфата (3,4). Кроме того, распознавание и катализ REases типа IIP тесно связаны. Таким образом, было трудно изменить специфичность распознавания при сохранении респектабельной активности (5).Для менее изученных REases типа IIS, распознавание последовательности-мишени и расщепление фосфодиэфирной связи осуществляются отдельными доменами (6–8). Такое разделение функций позволяет распознавать непалиндромы и расщеплять вне сайтов узнавания. Это также приводит к множеству механизмов распознавания ДНК и каталитических механизмов. Например, в AlwI (GGATC 4/5) замена домена расщепления ДНК доменом Nt.BstNBI (GAGTC 4/-) сохраняет специфичность распознавания AlwI, но превращает ее в в эндонуклеазу разрыва верхней цепи (NEase), показывая, что самого домена распознавания ДНК достаточно для специфического связывания с ДНК-мишенью (9). С точки зрения каталитического механизма, BfiI (ACTGGC 5/4) имеет каталитический центр типа фосфолипазы D и не требует ионов металлов для катализа (8). FokI (GGATG 9/13) имеет домен распознавания ДНК, который распознает GGATG, и домен расщепления ДНК, который несет канонический каталитический сайт PD-X n -DXK. Структурные и биохимические исследования показали, что временная димеризация FokI необходима для разрыва ds (7,10). Недавно сообщалось, что при смешивании FokI дикого типа (wt) с мутантом, который несет мутации важных ДНК-связывающих и каталитических остатков, гетеродимерный фермент специфически надрезает нижнюю цепь (11).
С точки зрения каталитического механизма, BfiI (ACTGGC 5/4) имеет каталитический центр типа фосфолипазы D и не требует ионов металлов для катализа (8). FokI (GGATG 9/13) имеет домен распознавания ДНК, который распознает GGATG, и домен расщепления ДНК, который несет канонический каталитический сайт PD-X n -DXK. Структурные и биохимические исследования показали, что временная димеризация FokI необходима для разрыва ds (7,10). Недавно сообщалось, что при смешивании FokI дикого типа (wt) с мутантом, который несет мутации важных ДНК-связывающих и каталитических остатков, гетеродимерный фермент специфически надрезает нижнюю цепь (11).
Ранее наша группа сообщила о двух REases типа IIS, которые содержат два независимых консервативных варианта каталитического мотива PD-X n -D/EXK. BtsI (GCAGTG 2/0) и BsrDI (GCAATG 2/0) представляют собой гетеродимерные REases типа IIS, которые распознают сходные последовательности из 6 оснований. Хотя каждая из их субъединиц несет каталитический сайт, только большая субъединица активна сама по себе и проявляет активность в отношении разрыва нижней цепи. Малая субъединица сама по себе не обладает активностью расщепления, но смешивание двух субъединиц приводит к активности расщепления ds. Когда каталитический сайт больших субъединиц BtsI и BsrDI мутирован, а затем комбинируется с их малыми субъединицами-партнерами дикого типа, оба фермента проявляют только активность надрыва верхней цепи (12). Сравнивая аминокислотные последовательности, мы обнаружили, что BsrI (ACTGG 1/-1) и Mva1269I/BsmI (GAATGC 1/-1) имеют сходные каталитические центры. Самое интересное, что в этих ферментах два каталитических сайта расположены на одном и том же полипептиде. Два каталитических сайта также работают на специфических цепях — мутация остатков в каждом из двух каталитических сайтов Mva1269I привела к появлению мутантов ДНК, специфичных для цепей. Кроме того, расщепление Mva1269I происходит в последовательном порядке: сначала расщепляется нижняя цепь, а затем верхняя (13).
Малая субъединица сама по себе не обладает активностью расщепления, но смешивание двух субъединиц приводит к активности расщепления ds. Когда каталитический сайт больших субъединиц BtsI и BsrDI мутирован, а затем комбинируется с их малыми субъединицами-партнерами дикого типа, оба фермента проявляют только активность надрыва верхней цепи (12). Сравнивая аминокислотные последовательности, мы обнаружили, что BsrI (ACTGG 1/-1) и Mva1269I/BsmI (GAATGC 1/-1) имеют сходные каталитические центры. Самое интересное, что в этих ферментах два каталитических сайта расположены на одном и том же полипептиде. Два каталитических сайта также работают на специфических цепях — мутация остатков в каждом из двух каталитических сайтов Mva1269I привела к появлению мутантов ДНК, специфичных для цепей. Кроме того, расщепление Mva1269I происходит в последовательном порядке: сначала расщепляется нижняя цепь, а затем верхняя (13).
Недавно NEases использовали для маркировки двухцепочечной ДНК путем ник-трансляции для обнаружения специфических последовательностей ДНК (14,15). В сочетании с визуализацией с высоким разрешением и растяжением ДНК сайты BbvCI были картированы на λ-ДНК с высокой скоростью и точностью (16). Такие методы картирования, вероятно, обеспечивают быструю идентификацию патогенов и мутаций, связанных с заболеванием, что, следовательно, ускоряет медицинскую диагностику. Однако ограниченное количество коммерчески доступных NEase ограничивает использование этих приложений.
В сочетании с визуализацией с высоким разрешением и растяжением ДНК сайты BbvCI были картированы на λ-ДНК с высокой скоростью и точностью (16). Такие методы картирования, вероятно, обеспечивают быструю идентификацию патогенов и мутаций, связанных с заболеванием, что, следовательно, ускоряет медицинскую диагностику. Однако ограниченное количество коммерчески доступных NEase ограничивает использование этих приложений.
Несколько родственных REases типа IIS (Mva1269I, BsmI, BsrI, BsrDI и BtsI) имеют два каталитических сайта: C b и C t . C b представляет собой каталитический сайт, специфичный для нижней цепи ДНК, тогда как C t специфичен для верхней цепи. Интересно, что BtsCI GGATG (2/0) и эти REases типа IIS расщепляют свои последовательности-мишени на 2 п.н. ниже динуклеотида верхней цепи TG, что приводит к 3′-выступам из 2 оснований (A). В BtsI и BsrDI C b представляет собой вариант канонического мотива REase PD-X 9. 0697 n -E-X n -QR, тогда как C t представляет собой канонический каталитический сайт PD-X n -EXK. Инактивация C b или C t путем мутагенеза создает варианты BtsI или BsrDI, которые надрезают только верхнюю или нижнюю цепь (12). Интересно, что в то время как два каталитических сайта расположены в разных субъединицах гетеродимерных BsrDI и BtsI, BsmI, BsrI и Mva1269I имеют оба каталитических сайта на одном полипептиде. Показано, что в Mva1269I, мутация остатков в каталитических сайтах приводила к вариантам никинга, которые связывают ДНК, содержащую сайт-мишень, так же сильно, как Mva1269I дикого типа (13).
0697 n -E-X n -QR, тогда как C t представляет собой канонический каталитический сайт PD-X n -EXK. Инактивация C b или C t путем мутагенеза создает варианты BtsI или BsrDI, которые надрезают только верхнюю или нижнюю цепь (12). Интересно, что в то время как два каталитических сайта расположены в разных субъединицах гетеродимерных BsrDI и BtsI, BsmI, BsrI и Mva1269I имеют оба каталитических сайта на одном полипептиде. Показано, что в Mva1269I, мутация остатков в каталитических сайтах приводила к вариантам никинга, которые связывают ДНК, содержащую сайт-мишень, так же сильно, как Mva1269I дикого типа (13).
Открыть в отдельном окне
( A ) Последовательности распознавания BtsCI и родственных REases выровнены по общим основаниям TG (подчеркнуто). Выделены общие 2-базовые 3′-выступы. Стрелками указаны сайты расщепления. ( B ) Выравнивание аминокислотных последовательностей двух каталитических сайтов BtsCI и родственных REases. Идентичные остатки показаны белым на черном фоне. Идентичность указывает процент идентичности аминокислотной последовательности между BtsCI и родственными REases. Последовательность ДНК системы BtsCI RM депонирована в GenBank под регистрационным номером {«type»:»entrez-нуклеотид»,»attrs»:{«text»:»GQ449683″,»term_id»:»270155117″,»term_text»:»GQ449683″}}GQ449683.
Идентичные остатки показаны белым на черном фоне. Идентичность указывает процент идентичности аминокислотной последовательности между BtsCI и родственными REases. Последовательность ДНК системы BtsCI RM депонирована в GenBank под регистрационным номером {«type»:»entrez-нуклеотид»,»attrs»:{«text»:»GQ449683″,»term_id»:»270155117″,»term_text»:»GQ449683″}}GQ449683.
В этой работе мы сообщаем о клонировании системы рестрикции-модификации (RM) BtsCI и о разработке специфичных для нити вариантов надрезов. , Наличие консервативных каталитических сайтов и создание вариантов разрывов путем мутации предполагаемых каталитических остатков подтверждает, что BtsCI принадлежит к группе REases Mva1269I/BsmI. рецессивные концы 3′ из 11 оснований, подходящие для мечения ДНК и лигирования, или 9-основание 5′ рецессивные концы для лигирования. Длинные выступы будут полезны для обнаружения и захвата ДНК на основе гибридизации.
Рестрикционные ферменты, ДНК-полимеразы, ДНК-субстраты, штаммы Escherichia coli (ER2683 и NEB Express), плазмидные векторы pBR322, pUC19, pACYC184, ДНК и белковые лестницы были получены от New England Biolabs Inc. BstF5I был приобретен у Sibenzyme.
BstF5I был приобретен у Sibenzyme.
Клонирование системы BtsCI R-M
ApoI, NlaIII или Sau3AI частично расщепленные геномные фрагменты из Bacillus thermosphaericus клонировали в расщепленную EcoRI, SphI или BamHI, обработанную CIP pUC19 с совместимыми когезионными концами, соответственно. Библиотеки плазмидной ДНК подвергали расщеплению BstF5I, изошизомера BtsCI. После заражения BstF5I выжившие (резистентные плазмиды) повторно трансформировали в E. coli . Индивидуальные трансформанты подвергали скринингу на устойчивость к расщеплению BstF5I, вставки из устойчивых клонов секвенировали и идентифицировали открытые рамки считывания (ORF). Полноразмерный ген эндонуклеазы BtsCI ( btsCIR) был получен методом обратной ПЦР и реклонирован в pUC19.
Штаммы для экспрессии белка
генов btsCIR и btsCIM лигировали с pUC19 и pACYC184 соответственно. E. coli ER2683 или NEB Express предварительно модифицировали с помощью pACYC- btsCIM с последующей трансформацией второй плазмидой pUC19- btsCIR для получения экспрессионного штамма: ER2683 [pACYC- btsCIM , pUC19-btsIR ]. Экспрессию BtsCI индуцировали добавлением 0,25 мМ IPTG к культуре клеток средней логарифмической фазы в среде LB в течение 3 ч при 37°C. Культуру собирали, а клеточный осадок хранили замороженным до лизиса для очистки белка.
Экспрессию BtsCI индуцировали добавлением 0,25 мМ IPTG к культуре клеток средней логарифмической фазы в среде LB в течение 3 ч при 37°C. Культуру собирали, а клеточный осадок хранили замороженным до лизиса для очистки белка.
Очистка белка
Осадок клеток из 1 л клеток, индуцированных IPTG, ресуспендировали в 40 мл 20 мМ трис-HCl, pH 7,5, 1 мМ ЭДТА и обрабатывали ультразвуком на льду. После центрифугирования при 15 000 g при 4°С в течение 30 мин надосадочную жидкость нагревали при 60°С в течение 30 мин. денатурированный 9Белок 0697 E. coli удаляли центрифугированием при 20 000 g в течение 30 мин при 4°С. Супернатант подвергали колоночной хроматографии через Heparin HyperD® M (Pall) и Q-Sepharose™ (GE Life Sciences) с линейными градиентами NaCl (50 мМ–1 М). Пиковые фракции анализировали на активность расщепления или образования разрывов и объединяли (подробная процедура очистки доступна по запросу).
Сайт-направленный мутагенез и скрининг вариантов разрывов
Мутации вводили с помощью варианта метода обратной ПЦР с использованием pUC19- btsCI R в качестве шаблона (17). Новые конструкции секвенировали для подтверждения желаемых мутаций.
Новые конструкции секвенировали для подтверждения желаемых мутаций.
Определение олигомерного состояния
Молекулярную массу вариантов BtsCI определяли в экспериментах по равновесной гель-фильтрации (18) с использованием системы жидкостной хроматографии белков AKTA FPLC с колонкой Superdex 200 10/300 GL (GE Healthcare). Колонку уравновешивали буфером, содержащим 20 мМ Трис-HCl, рН 7,5, 200 мМ NaCl. Калибровочную кривую строили с использованием набора для калибровки с высокой молекулярной массой (GE Healthcare).
Анализ активности расщепления/разрыва и идентификация расщепленных цепей и сайтов расщепления
Активность расщепления/разрыва экстрактов мутантных клеток BtsCI или очищенных ферментов анализировали на 0,5 мкг ДНК pUC19 в NEBuffer 4 при 50°C в течение 1 часа. Продукты расщепления/разрыва анализировали с помощью гель-электрофореза с использованием 1,5% агарозных гелей. На pUC19 имеется пять сайтов BtsCI. Следовательно, полное расщепление приводит к размерам фрагментов 181, 244, 287, 613 и 1361 п. н., как и предсказывает NEBcutter (19).). Поскольку ближайшие соседние сайты находятся на расстоянии 181 п.н. друг от друга, линеаризованная ДНК не должна наблюдаться в условиях электрофореза в агарозном геле, если сайты преимущественно разорваны. Для идентификации расщепленных цепей и сайтов расщепления ДНК с разрывами очищали в геле и подвергали секвенированию по Сэнгеру (набор для циклического секвенирования BigDye® Terminator Cycle Sequencing Kit, Applied Biosystems).
н., как и предсказывает NEBcutter (19).). Поскольку ближайшие соседние сайты находятся на расстоянии 181 п.н. друг от друга, линеаризованная ДНК не должна наблюдаться в условиях электрофореза в агарозном геле, если сайты преимущественно разорваны. Для идентификации расщепленных цепей и сайтов расщепления ДНК с разрывами очищали в геле и подвергали секвенированию по Сэнгеру (набор для циклического секвенирования BigDye® Terminator Cycle Sequencing Kit, Applied Biosystems).
Получение специфических выступов и обнаружение
Плазмида pUC19 (0,25 мкг) расщепляли с помощью FokI (5 единиц) в NEBuffer 4 при 37°C в течение 30 мин. Для создания 11-базового выступа 5 футов или 9-основание 3′-выступ, пять единиц Nt.BtsCI или Nb.BtsCI добавляли к расщепленным FokI реакциям и инкубировали при 50°C в течение 1 часа. Расщепленный продукт лигировали с дуплексным олигонуклеотидом (длиной ~100 п.н.), содержащим комплементарный выступ, при комнатной температуре в течение 2 часов. Лигированный продукт обнаруживали с помощью ПЦР, где один из праймеров отжигается с дуплексным олигонуклеотидом, а другой — с pUC19 рядом с сайтом FokI. Продукт ПЦР подвергали электрофорезу в агарозном геле.
Продукт ПЦР подвергали электрофорезу в агарозном геле.
Мечение ДНК путем заполнения концов с помощью Cy3-dNTP
Последовательности ДНК, содержащие два сайта узнавания BtsCI, амплифицировали из pUC19 с использованием мастер-микса LongAmp Taq 2X и расщепляли указанными эндонуклеазами. Расщепление останавливали добавлением ЭДТА до конечной концентрации 20 мМ. Продукт ПЦР (0,1 мкг) метили Cy3-dNTP (PerkinElmer) фрагментом Кленова E. coli Pol I при комнатной температуре в течение 15 мин, а затем анализировали с помощью 6% PAGE в 1 × TBE. Меченую ДНК визуализировали с помощью лазерного сканера Typhoon 9400 (GE Life Sciences, возбуждение — 532 нм, эмиссия — 580 нм). Тотальную ДНК визуализировали с помощью УФ-освещения после окрашивания бромистым этидием (2 мкг/мл).
Клонирование системы BtsCI R-M
Стратегию селекции метилазой (20) использовали для клонирования гена модификации (M) BtsCI и части гена рестрикции (R), а обратную ПЦР-прогулку использовали для получения полноразмерного гена R . В системе BtsCI RM были обнаружены два гена, btsCIM (2064 п.н.) и btsCIR (1398 п.н.). Мы субклонировали гена btsCIM в pACYC184 для получения предварительно модифицированного хозяина (ER2683). Плазмида pACYC- btsCIM , очищенная от Было подтверждено, что штамм E. coli устойчив к расщеплению BtsCI (модификация сайтов BtsCI метилазой BtsCI придает плазмиде устойчивость к расщеплению BtsCI). Ген btsCIR , содержащийся в ПЦР-фрагменте, затем лигировали в pUC19 и вводили предварительно модифицированному хозяину. Активность BtsCI REase была обнаружена в экстрактах клеток, индуцированных IPTG (данные не показаны).
В системе BtsCI RM были обнаружены два гена, btsCIM (2064 п.н.) и btsCIR (1398 п.н.). Мы субклонировали гена btsCIM в pACYC184 для получения предварительно модифицированного хозяина (ER2683). Плазмида pACYC- btsCIM , очищенная от Было подтверждено, что штамм E. coli устойчив к расщеплению BtsCI (модификация сайтов BtsCI метилазой BtsCI придает плазмиде устойчивость к расщеплению BtsCI). Ген btsCIR , содержащийся в ПЦР-фрагменте, затем лигировали в pUC19 и вводили предварительно модифицированному хозяину. Активность BtsCI REase была обнаружена в экстрактах клеток, индуцированных IPTG (данные не показаны).
Поиск BLASTP показал, что M.BtsCI имеет высокое сходство последовательностей с изошизомерами M.FokI (идентичность последовательностей 62%/сходство последовательностей 75%) и M.StsI (идентичность последовательностей 48%/сходство последовательностей 65%) (данные не показаны) . N-конец M.BtsCI подобен M3.BstF5I, а C-конец M. BtsCI подобен M2.BstF5I, что позволяет предположить, что M.BtsCI представляет собой слияние двух функциональных метилаз, которые модифицируют каждую из двух цепей. Аминокислотные последовательности BtsCI и FokI REases не имеют значительного сходства. Однако два гомолога эндонуклеаз BtsCI (предполагаемых эндонуклеаз) были обнаружены из двух секвенированных микробных геномов (см. ниже).
BtsCI подобен M2.BstF5I, что позволяет предположить, что M.BtsCI представляет собой слияние двух функциональных метилаз, которые модифицируют каждую из двух цепей. Аминокислотные последовательности BtsCI и FokI REases не имеют значительного сходства. Однако два гомолога эндонуклеаз BtsCI (предполагаемых эндонуклеаз) были обнаружены из двух секвенированных микробных геномов (см. ниже).
Каталитические мотивы BtsCI
Выравнивание последовательностей между BtsCI и аналогичными ферментами показывает, что BtsCI содержит эти два каталитических сайта, подобных Mva1269I/BsmI, хотя общая идентичность последовательностей низкая (B). Мотив SD-X 6 -E-X 14 -QR был обнаружен вблизи N-конца BtsCI, а мотив, подобный C t REases типа Mva1269I/BsmI, был обнаружен вблизи С-конца. . Интересно, что хотя мотив C t является каноническим PD-X n -EXK для ферментов, подобных Mva1269I/BsmI, мотив C t в BtsCI более похож на BamHI и содержит PD-X n n -EXE (E вместо K) (B). Это побудило нас проверить каталитические мотивы и сконструировать специфичные для нитей варианты BtsCI с надрезами.
Это побудило нас проверить каталитические мотивы и сконструировать специфичные для нитей варианты BtsCI с надрезами.
Генерация никинговых вариантов BtsCI
Одиночные мутации D121A, E128A и R145A были введены в мотив C b в BtsCI на основе аналогичных мутаций D58A, E72A и R86A в большой субъединице BtsI (B) (12). показывает, что эти мутанты утратили большую часть своей активности по расщеплению двухцепочечной ДНК и только порезали pUC19.CATCC (данные не показаны). Этот результат подтверждает предположение, что N-концевой каталитический сайт отвечает за расщепление нижней цепи (C b ), а C-концевой сайт отвечает за расщепление верхней цепи (C t ) для Mva1268I/BsmI-подобного РАС.
Открыть в отдельном окне
Активность ДНК-никинга мутантов BtsCI. Двукратные серийные разведения осветленных клеточных экстрактов культур E. coli , экспрессирующих указанные мутанты BtsCI, инкубировали с 0,5 мкг pUC19.как описано в разделе «Материалы и методы». Продукты расщепления анализировали на 1% агарозном геле. ОС, открытый круг; СК, сверхскрученный; − без спайности; +, pUC19 взломан Nt.BsmAI.
Продукты расщепления анализировали на 1% агарозном геле. ОС, открытый круг; СК, сверхскрученный; − без спайности; +, pUC19 взломан Nt.BsmAI.
Улучшение вариантов надрезов BtsCI
Хотя мутанты D388A и E403A/E405A демонстрируют сильную активность надрезов нижней цепи, оба они по-прежнему демонстрируют значительное расщепление двухцепочечной ДНК при более высоких концентрациях фермента (данные не показаны). Точно так же мутанты с разрывом верхней цепи D121A, E128A, R145A и D121A/E128A также обладают обнаруживаемой активностью по расщеплению двухцепочечной ДНК (данные не показаны). Для минимизации активности расщепления двухцепочечной ДНК проводили дальнейший мутагенез.
Сначала D388 C t был мутирован на противоположно заряженный аминокислотный остаток Lys (K). Но D388K был неактивен (данные не приведены). Когда аминокислотные замены D388A, E403A и E405A были объединены, тройной мутант D388A/E403A/E405A демонстрировал высокий уровень экспрессии белка, высокую активность в отношении разрыва нижней цепи и минимальную активность расщепления двухцепочечной ДНК (), предполагая, что комбинация трех аминокислот замены полностью инактивирует каталитический центр верхней цепи.
Открыть в отдельном окне
Активность разрыва ДНК варианта BtsCI с разрывом нижней цепи. Двукратные серийные разведения неочищенного экстракта варианта BtsCI с порезами (D388A/E403A/E405A) инкубировали с pUC19, как описано в разделе «Материалы и методы». Продукты расщепления анализировали на 1% агарозном геле. ОС, открытый круг; СК, сверхспиральный.
В случае надрыва верхней цепи объединение трех мутаций, вызывающих активность надрезов верхней цепи (тройной мутант D121A/E128A/R145A), не устраняло активность расщепления двухцепочечной ДНК (данные не показаны). Отмена заряда для C 9Каталитические остатки 0808 b (двойной мутант D121K/E128R) имели очень низкий уровень экспрессии и более низкую активность в отношении разрыва по сравнению с двойным мутантом D121A/E128A (данные не показаны). Однако когда E128 заменяли на Phe (F), полученный мутант E128F обладал высокой активностью разрыва и низкой активностью расщепления двухцепочечной ДНК (1). Возможно, объемная ароматическая боковая цепь Phe обеспечивает более эффективное вмешательство в реакцию расщепления C b . Мы пришли к выводу, что есть как минимум две стратегии улучшения вариантов взлома. Первый — путем комбинации множественных аминокислотных замен из вариантов с несовершенными разрывами (конструкция двойных или тройных мутантов). Вторая стратегия заключается в проведении мутагенеза с насыщением определенного аминокислотного остатка и поиске улучшенных свойств фермента.
Мы пришли к выводу, что есть как минимум две стратегии улучшения вариантов взлома. Первый — путем комбинации множественных аминокислотных замен из вариантов с несовершенными разрывами (конструкция двойных или тройных мутантов). Вторая стратегия заключается в проведении мутагенеза с насыщением определенного аминокислотного остатка и поиске улучшенных свойств фермента.
Открыть в отдельном окне
Активность разрыва ДНК варианта BtsCI с разрывом верхней цепи. Двукратные серийные разведения клеточного экстракта BtsCI никирующего варианта E128F инкубировали с pUC19, как описано в разделе «Материалы и методы». Продукты расщепления анализировали на 1% агарозном геле. ОС, открытый круг; СК, сверхспиральный.
Специфичность сайта-мишени и разрывной нити с помощью вариантов BtsCI E128F и D388A/E403A/E405A была подтверждена секвенированием по методу Sanger «run-off» (). Эти два мутанта были обозначены как Nt.BtsCI и Nb.BtsCI соответственно и использовались в следующих экспериментах.
Открыть в отдельном окне
Вывод секвенирования надрезанных изделий по вариантам надрезов БЦИ. Продукт расщепления мутантов E128F ( A ) и D388A/E403A/E405A ( B ) очищали на геле и подвергали секвенированию обеих цепей по Сэнгеру. Последовательность распознавания выделена жирным шрифтом, а сайт расщепления указан стрелками. Дополнительный пик А в конце последовательности стока был добавлен независимой от матрицы концевой трансферазной активностью ДНК-полимеразы Taq во время секвенирования. Дополнительный пик А и падение пикового сигнала указывают на место разрыва (расщепленный шаблон).
BtsCI является мономерным белком
Wt-BtsCI, Nt.BtsCI и Nb.BtsCI были очищены до степени чистоты более 95% (A), и для определения состояния олигомеризации этих белков в растворе была использована гель-фильтрация. Для каждого белка был получен один пик, и его элюировали с кажущейся молекулярной массой ~40–45 кДа, что соответствует мономерному белку (B). Специфическое расщепление ДНК (для фермента дикого типа) или активность разрыва (для вариантов разрыва) присутствовали во фракциях пика (данные не показаны).
Специфическое расщепление ДНК (для фермента дикого типа) или активность разрыва (для вариантов разрыва) присутствовали во фракциях пика (данные не показаны).
Открыть в отдельном окне
( A ) SDS-PAGE анализ BtsCI дикого типа и двух вариантов разрыва. ( B ) Определение состояния олигомеризации вариантов дикого типа BtsCI, Nt и Nb с никотинированием с помощью гель-фильтрационной хроматографии. Объем элюции белкового стандарта наносили на график в зависимости от соответствующей молекулярной массы (в логарифмической шкале). Стандартные белки (открытые квадраты): овальбумин, 44 кДа; кональбумин, 75 кДа; Альдолаза, 158 кДа; ферритин, 440 кДа; Тиреоглобулин, 669кДа. R 2 стандартной кривой составляет 0,9937. Белки BtsCI (черный квадрат) были расположены на калибровочной кривой, и соответствующие молекулярные массы были рассчитаны с использованием уравнения калибровочной кривой: объем элюции = -4,9763 (логарифм молекулярной массы) + 39,39.
Создание длинных «липких» концов путем комбинации расщепления FokI и N.BtsCI
Варианты с разрывами BtsCI могут надрезать верхнюю или нижнюю цепь ДНК, расщепленной FokI, и генерировать относительно длинные выступающие части. ДНК, разрезанная с помощью FokI/Nt.BtsCI, будет иметь 3′-рецессивный конец из 11 оснований (5′-выступ), тогда как расщепление FokI/Nb.BtsCI может генерировать 9-основание 5′ рецессивный конец (3′-выступ) (А). Для создания длинных выступов необходимо последовательное пищеварение. Отчасти это связано с тем, что FokI и BtsCI имеют разные оптимальные температуры для активности расщепления. Мы также обнаружили, что совместная инкубация FokI и N.BtsCI при 37°C приводила к снижению активности FokI (данные не показаны), возможно, из-за конкуренции между FokI и BtsCI за одни и те же сайты узнавания.
Открыть в отдельном окне
( A ) Генерация свесов. Указаны сайты расщепления для BtsCI, FokI и комбинации FokI/Nt.BtsCI и FokI/Nb. BtsCI. Для BtsCI генерируется 2-нуклеотидный 5′-рецессивный конец. Для FokI генерируется рецессивный 3′-конец из 4 нуклеотидов. Для FokI/Nt.BtsCI генерируется рецессивный 3′-конец из 11 нуклеотидов. Для ФокИ/Нб.БцКИ 9Генерируется рецессивный 5′-конец -nt. Серые стрелки, сайт расщепления верхней цепи BtsCI; белые стрелки — сайт расщепления нижней цепи BtsCI; черные стрелки — сайты расщепления FokI. ( B ) Отжиг олигонуклеотидов до длинных выступов для ПЦР. Плазмида pUC19 была расщеплена указанным ферментом (ферментами), а затем лигирована с олигонуклеотидом длиной 100 п.н., как описано в разделе «Материалы и методы». Продукты лигирования использовали в качестве матрицы для ПЦР, которая специфически выявляла лигированную ДНК. Только продукт расщепления FokI/Nt.BtsCI может быть отожжен до 11-нуклеотидного 3′-рецессивного концевого олигонуклеотида (левая панель), тогда как только продукт расщепления FokI/Nb.BtsCI может быть отожжен до 9-nt 5′ рецессивный концевой олигонуклеотид (правая панель).
BtsCI. Для BtsCI генерируется 2-нуклеотидный 5′-рецессивный конец. Для FokI генерируется рецессивный 3′-конец из 4 нуклеотидов. Для FokI/Nt.BtsCI генерируется рецессивный 3′-конец из 11 нуклеотидов. Для ФокИ/Нб.БцКИ 9Генерируется рецессивный 5′-конец -nt. Серые стрелки, сайт расщепления верхней цепи BtsCI; белые стрелки — сайт расщепления нижней цепи BtsCI; черные стрелки — сайты расщепления FokI. ( B ) Отжиг олигонуклеотидов до длинных выступов для ПЦР. Плазмида pUC19 была расщеплена указанным ферментом (ферментами), а затем лигирована с олигонуклеотидом длиной 100 п.н., как описано в разделе «Материалы и методы». Продукты лигирования использовали в качестве матрицы для ПЦР, которая специфически выявляла лигированную ДНК. Только продукт расщепления FokI/Nt.BtsCI может быть отожжен до 11-нуклеотидного 3′-рецессивного концевого олигонуклеотида (левая панель), тогда как только продукт расщепления FokI/Nb.BtsCI может быть отожжен до 9-nt 5′ рецессивный концевой олигонуклеотид (правая панель).
Чтобы подтвердить образование длинных выступающих частей, расщепленную FokI/N.BtsCI pUC19 лигировали с дуплексным олигонуклеотидом, который содержит комплементарный выступающий конец, с использованием ДНК-лигазы Т4. Лигированный продукт затем обнаруживали с помощью ПЦР, где один из праймеров отжигался с олигонуклеотидным адаптером, а другой отжигался с последовательностью pUC19. Специфический продукт ПЦР (около 200 п.н.) можно было наблюдать только в субстрате, расщепленном FokI/Nt.BtsCI или FokI/Nb.BtsCI, что указывает на то, что расщепление является специфическим и были получены ожидаемые выступы (B).
Мечение ДНК путем заполнения 11-нуклеотидного 3′-рецессивного конца
11-нуклеотидный 3′-рецессивный конец, образованный FokI/Nt.BtsCI, можно использовать для мечения за счет удлиняющей активности ДНК-полимераз. Фрагмент ДНК длиной 406 п.н., содержащий два сайта BtsCI, амплифицировали с помощью ПЦР и расщепляли FokI, FokI/Nt.BtsCI или FokI/Wt-BtsCI. Затем продукты расщепления удлиняли с использованием фрагмента Кленова в присутствии Cy3-dNTP с последующим анализом с помощью 6% PAGE. Нижняя панель иллюстрирует ожидаемые продукты расщепления и рецессивные 3′-концы, которые можно пометить путем заполнения концов. Продукт расщепления FokI/Nt.BtsCI имеет сильный флуоресцентный сигнал в нижней полосе дублета (приблизительно 176 п.н.) после реакции удлинения, что указывает на эффективное мечение выступающего конца. Более слабый сигнал был обнаружен в продукте расщепления FokI, поскольку FokI генерировал более короткий выступ из 4 оснований. С другой стороны, 5′-выступ из 2 оснований, генерируемый BtsCI, не был помечен.
Нижняя панель иллюстрирует ожидаемые продукты расщепления и рецессивные 3′-концы, которые можно пометить путем заполнения концов. Продукт расщепления FokI/Nt.BtsCI имеет сильный флуоресцентный сигнал в нижней полосе дублета (приблизительно 176 п.н.) после реакции удлинения, что указывает на эффективное мечение выступающего конца. Более слабый сигнал был обнаружен в продукте расщепления FokI, поскольку FokI генерировал более короткий выступ из 4 оснований. С другой стороны, 5′-выступ из 2 оснований, генерируемый BtsCI, не был помечен.
Открыть в отдельном окне
Мечение ДНК путем заполнения 11-нуклеотидного 3′-рецессивного конца. Рецессивный 3′-конец из 11 нуклеотидов был получен с помощью FokI/Nt.BtsCI. Сильный флуоресцентный сигнал обнаружен на меньшем фрагменте ДНК (нижняя полоса), расщепленном FokI/Nt.BtsCI. Ожидаемые продукты расщепления и рецессивные 3′-концы, которые могут быть помечены заполнением концов, показаны на нижней панели. Зеленые блоки, область флуоресцентной маркировки; серые стрелки, сайт расщепления верхней цепи BtsCI; белые стрелки — сайт расщепления нижней цепи BtsCI; черные стрелки — сайты расщепления FokI.
REases типа IIS с двумя каталитическими сайтами
REases типа IIS представляют собой интересную группу REases с точки зрения разнообразия способов распознавания целевой последовательности и каталитического механизма. В отличие от наиболее распространенных REases типа IIP, которые имеют один каталитический сайт на молекулу, содержащую остатки распознавания ДНК, REases типа IIS неизменно имеют отдельные домены распознавания и расщепления ДНК (12, 21–23). Это разделение функций приводит к множеству механизмов расщепления ДНК. В этой работе мы сосредоточимся на BtsCI, который принадлежит Mva1269.I/BsmI группа REases, которые несут два разных каталитических сайта на одном и том же полипептиде. Два каталитических сайта аналогичны каноническому PD-X n -D/EXK (X — любой аминокислотный остаток), но расходятся. В BtsCI каталитический сайт разрыва нижней цепи (C b ) соответствует мотиву SD-X 6 -E-X 14 -QR Mva1269I/BsmI-подобных REases, расположенному вблизи N-конца (B) . Интересно, что каталитический сайт верхней цепи C t является каноническим PD-X n -EXK для Mva1269I/BsmI-подобных ферментов, но становится BamHI-подобным PD-X n -EXE в BtsCI (B).
Интересно, что каталитический сайт верхней цепи C t является каноническим PD-X n -EXK для Mva1269I/BsmI-подобных ферментов, но становится BamHI-подобным PD-X n -EXE в BtsCI (B).
BtsI и BsrDI также содержат эти два каталитических центра, но они расположены в двух отдельных субъединицах. Димеризация необходима для BtsI и BsrDI, чтобы сделать разрывы двухцепочечной ДНК. Ранее мы показали, что мутации остатков каждого из каталитических сайтов приводят к специфичным для нитей вариантам надрезов для BtsI и BsrDI (12). Аналогичные результаты были также получены для Mva1269.я (13). Расщепление/надрез каждой цепи с помощью Mva1269I является последовательным (т. е. сначала надрезается нижняя цепь, ДНК с надрезом нижней цепи является подходящим субстратом для надрезания верхней цепи. Фермент надреза верхней цепи имеет очень низкую активность).
В дополнение к семейству Mva1269I/BsmI/BsrI/BtsCI несколько REases типа IIS также содержат отдельные каталитические сайты, каждый из которых отвечает за расщепление определенной цепи ДНК. Системы BstNBI и BspD6I (GAGTC 4/6), о которых сообщают две независимые группы, идентичны по аминокислотным последовательностям и первоначально были известны как естественные никирующие ферменты (24, 25). Когда продукт гена соседней ORF BspD6I очищают и добавляют к ферменту, создающему разрывы BspD6I, фермент расщепляет обе нити (26). Подобно BtsI и BsrDI, малая субъединица BspD6I (генный продукт соседней ORF) сама по себе не обладает активностью расщепления. Более интересно, что малая субъединица в высокой степени гомологична С-концу никирующего фермента BspD6I. Остается провести работу по мутагенезу, чтобы проверить, приводит ли мутация каталитического сайта малой субъединицы к мутантам с разрывом, которые разрывают исключительно противоположную цепь.
Системы BstNBI и BspD6I (GAGTC 4/6), о которых сообщают две независимые группы, идентичны по аминокислотным последовательностям и первоначально были известны как естественные никирующие ферменты (24, 25). Когда продукт гена соседней ORF BspD6I очищают и добавляют к ферменту, создающему разрывы BspD6I, фермент расщепляет обе нити (26). Подобно BtsI и BsrDI, малая субъединица BspD6I (генный продукт соседней ORF) сама по себе не обладает активностью расщепления. Более интересно, что малая субъединица в высокой степени гомологична С-концу никирующего фермента BspD6I. Остается провести работу по мутагенезу, чтобы проверить, приводит ли мутация каталитического сайта малой субъединицы к мутантам с разрывом, которые разрывают исключительно противоположную цепь.
Эндонуклеаза BtsCI имеет очень небольшое сходство аминокислотной последовательности с неошизомерами FokI и StsI. Однако он показывает 33% идентичность аминокислотной последовательности с двумя консервативными гипотетическими белками Cup_ORF1489 (463 а. о., GI:57015558) и Nfl_ORF1032 (472 а.о., GI:241320910), обнаруженными в Campylobacter upsaliensis RM3195 и Neisseria flavescens 16498SK 16498 SK 16498 SK 16498 SK 16498 SK 16498 SK 16498 SK 16498 SK 16498 . Оба белка сопровождаются двумя ДНК-метилазами (27,28). M1.Cup_ORF1489 и M2. Nfl_ORF1032 демонстрирует очень высокую гомологию аминокислотной последовательности с N-концевым доменом M.BtsCI, а аминокислотная последовательность С-концевого домена M.BtsCI гомологична M2.Cup_ORF1489.и М1. Nfl_ORF1032 (данные не показаны). Предполагается, что Cup_ORF1489 и Nfl_ORF1032 также несут два каталитических сайта. Неканонический мотив S(K)D(E)-X 6 -E-X 14 -QR больше похож на BtsCI, тогда как канонический мотив PD-X n -EXK больше похож на BtsCI. больше похож на Mva1269I/BsmI (B). Весьма вероятно, что продукты генов Cup_ORF1489 и Nfl_ORF1032 представляют собой изошизомеры BtsCI, которые узнают и расщепляют последовательность GGATG.
о., GI:57015558) и Nfl_ORF1032 (472 а.о., GI:241320910), обнаруженными в Campylobacter upsaliensis RM3195 и Neisseria flavescens 16498SK 16498 SK 16498 SK 16498 SK 16498 SK 16498 SK 16498 SK 16498 SK 16498 . Оба белка сопровождаются двумя ДНК-метилазами (27,28). M1.Cup_ORF1489 и M2. Nfl_ORF1032 демонстрирует очень высокую гомологию аминокислотной последовательности с N-концевым доменом M.BtsCI, а аминокислотная последовательность С-концевого домена M.BtsCI гомологична M2.Cup_ORF1489.и М1. Nfl_ORF1032 (данные не показаны). Предполагается, что Cup_ORF1489 и Nfl_ORF1032 также несут два каталитических сайта. Неканонический мотив S(K)D(E)-X 6 -E-X 14 -QR больше похож на BtsCI, тогда как канонический мотив PD-X n -EXK больше похож на BtsCI. больше похож на Mva1269I/BsmI (B). Весьма вероятно, что продукты генов Cup_ORF1489 и Nfl_ORF1032 представляют собой изошизомеры BtsCI, которые узнают и расщепляют последовательность GGATG.
Применение длинных выступов
FokI и BtsCI являются неошизомерами, которые расщепляются на различном расстоянии ниже по течению от GGATG. Мы воспользовались этим свойством и получили 11-нуклеотидные 3′-рецессивные концы или 9-нуклеотидные 5′-рецессивные концы путем последовательного расщепления с помощью FokI и Nt.BtsCI или Nb.BtsCI соответственно. Мы показали, что оба конца могут быть лигированы с комплементарными линкерами (B). Рецессивные 3′-концы из 11 нуклеотидов могут быть помечены фрагментом Кленова с использованием флуоресцентно-меченых dNTP (4). Длинные свесы могут использоваться в самых разных областях. Биотинилированные олигонуклеотиды, комплементарные выступам, можно использовать для извлечения специфических фрагментов ДНК с использованием гранул стрептавидина для обнаружения и осаждения комплексов белок-ДНК в приложениях с высокой пропускной способностью, таких как SAGE. Рецессивные 3′-концы из 11 оснований содержат больше нуклеотидов для высокоуровневой маркировки флуоресцентно-мечеными нуклеотидами, чем рецессивные концы из 4 оснований 3′, образованные с использованием одной REase (11). Относительно частая распознаваемая последовательность из 5 оснований FokI/BtsCI также позволяет быстро обнаруживать полиморфизмы ДНК посредством прямого и высокоуровневого мечения посредством заполнения концов.
Мы воспользовались этим свойством и получили 11-нуклеотидные 3′-рецессивные концы или 9-нуклеотидные 5′-рецессивные концы путем последовательного расщепления с помощью FokI и Nt.BtsCI или Nb.BtsCI соответственно. Мы показали, что оба конца могут быть лигированы с комплементарными линкерами (B). Рецессивные 3′-концы из 11 нуклеотидов могут быть помечены фрагментом Кленова с использованием флуоресцентно-меченых dNTP (4). Длинные свесы могут использоваться в самых разных областях. Биотинилированные олигонуклеотиды, комплементарные выступам, можно использовать для извлечения специфических фрагментов ДНК с использованием гранул стрептавидина для обнаружения и осаждения комплексов белок-ДНК в приложениях с высокой пропускной способностью, таких как SAGE. Рецессивные 3′-концы из 11 оснований содержат больше нуклеотидов для высокоуровневой маркировки флуоресцентно-мечеными нуклеотидами, чем рецессивные концы из 4 оснований 3′, образованные с использованием одной REase (11). Относительно частая распознаваемая последовательность из 5 оснований FokI/BtsCI также позволяет быстро обнаруживать полиморфизмы ДНК посредством прямого и высокоуровневого мечения посредством заполнения концов. 9, каталог NEB 2009/10, стр. 79). Терминазы некоторых бактериальных фагов также образуют длинные выступы. Однако фаговые терминазы обычно обладают низкой специфической активностью и требуют для расщепления АТФ и конкатемерной ДНК (29). С другой стороны, описанный здесь метод является высокоэффективным и специфичным для сайтов FokI/BtsCI.
9, каталог NEB 2009/10, стр. 79). Терминазы некоторых бактериальных фагов также образуют длинные выступы. Однако фаговые терминазы обычно обладают низкой специфической активностью и требуют для расщепления АТФ и конкатемерной ДНК (29). С другой стороны, описанный здесь метод является высокоэффективным и специфичным для сайтов FokI/BtsCI.
Недавно Sanders и коллеги (11) сообщили о генерации специфичной для нити активности FokI путем специфического инактивирования ДНК-связывания и/или активности расщепления ДНК. В их схеме расщепление смесью FokI дикого типа и мутантов с дефицитом связывания ДНК и дефицитом расщепления вызывает активность расщепления нижней цепи, тогда как расщепление с использованием комбинации варианта FokI с дефицитом связывания ДНК и дефицита расщепления Вариант FokI вызывает активность надрезов верхней нити. Хотя в последнем случае расщепляется исключительно верхняя цепь, в первом случае верхняя цепь также расщепляется в значительной степени, поскольку присутствие FokI дикого типа приводит к гомодимеризации, которая расщепляет обе цепи. В принципе, расщепление гетеродимером FokI с нижним соединением, за которым следует Nt.BtsCI, будет генерировать фрагменты ДНК, каждый из которых содержит 3′-рецессивный конец из 11 оснований, способный к сборке фрагментов ДНК без лигазы.
В принципе, расщепление гетеродимером FokI с нижним соединением, за которым следует Nt.BtsCI, будет генерировать фрагменты ДНК, каждый из которых содержит 3′-рецессивный конец из 11 оснований, способный к сборке фрагментов ДНК без лигазы.
BtsCI и FokI демонстрируют очень небольшую гомологию аминокислотных последовательностей, несмотря на их идентичную последовательность узнавания ДНК. BtsCI объединяет BsrI, BsmI, Mva1269I, BbvCI и гетеродимерные BtsI, BsrDI и BstNBI/BspD6I в группу REases типа IIS, которые содержат два каталитических сайта, каждый из которых действует на специфическую цепь субстрата двухцепочечной ДНК. Были созданы варианты BtsCI, которые разрезают определенную цепь целевого сайта. Используя двойное расщепление FokI/N.BtsCI, можно получить рецессивный 3′-конец из 11 оснований или 9рецессивный конец 5′ основания для обнаружения/захвата ДНК на основе гибридизации и мечения концов ДНК.
ФИНАНСИРОВАНИЕ
Финансирование платы за открытый доступ: New England Biolabs.
Заявление о конфликте интересов . Ни один не заявил.
Последовательность ДНК системы рестрикции-модификации BtsCI депонирована в GenBank под инвентарным номером {«type»:»entrez-нуклеотид»,»attrs»:{«text»:»GQ449683″,»term_id»:»270155117 «,»term_text»:»GQ449683″}}GQ449683.
Авторы благодарят Рича Робертса, Джеффа Уилсона и Билла Джека за критические комментарии и обсуждение; X. Пан для предоставления нативного штамма 9, продуцирующего BtsCI.0697 Bacillus thermosphaericus ; Дону Комбу и Джиму Элларду за финансовую поддержку.
1. Пингоуд А., Йелч А. Структура и функция эндонуклеаз рестрикции II типа. Нуклеиновые Кислоты Res. 2001; 29: 3705–3727. [Бесплатная статья PMC] [PubMed] [Google Scholar]
2. Pingoud A, Fuxreiter M, Pingoud V, Wende W. Эндонуклеазы рестрикции II типа: структура и механизм. Ячейка Мол. Жизнь наук. 2005; 62: 685–707. [PubMed] [Google Scholar]
3. Ньюман М., Стшелецка Т., Дорнер Л.Ф., Шильдкраут И., Аггарвал А. К. Структура эндонуклеазы Bam HI, связанной с ДНК: частичная укладка и разворачивание при связывании ДНК. Наука. 1995;269:656–663. [PubMed] [Google Scholar]
К. Структура эндонуклеазы Bam HI, связанной с ДНК: частичная укладка и разворачивание при связывании ДНК. Наука. 1995;269:656–663. [PubMed] [Google Scholar]
4. Ньюман М., Стшелецка Т., Дорнер Л.Ф., Шильдкраут И., Аггарвал А.К. Структура эндонуклеазы рестрикции BamHI и ее связь с EcoRI. Природа. 1994; 368: 660–664. [PubMed] [Google Scholar]
5. Townson SA, Samuelson JC, Xu SY, Aggarwal AK. Последствия переключения специфичности рестрикционных ферментов со структуры BstYI, связанной с последовательностью ДНК BglII. Структура. 2005; 13: 791–801. [PubMed] [Google Scholar]
6. Yonezawa A, Sugiura Y. Способ связывания ДНК рестрикционной эндонуклеазы класса IIS FokI, выявленный с помощью анализа следов ДНК. Биохим. Биофиз. Акта. 1994;1219:369–379. [PubMed] [Google Scholar]
7. Вах Д.А., Битинайте Дж., Шильдкраут И., Аггарвал А.К. Структура FokI имеет значение для расщепления ДНК. проц. Натл акад. наук США. 1998; 95:10564–10569. [Статья бесплатно PMC] [PubMed] [Google Scholar]
8. Гразулис С., Манакова Э., Россле М., Бохтлер М., Тамулайтиен Г., Хубер Р., Сикснис В. Структура металл-независимого рестриктазы BfiI обнаруживает слияние специфический ДНК-связывающий домен с неспецифической нуклеазой. проц. Натл акад. наук США. 2005;102:15797–15802. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Гразулис С., Манакова Э., Россле М., Бохтлер М., Тамулайтиен Г., Хубер Р., Сикснис В. Структура металл-независимого рестриктазы BfiI обнаруживает слияние специфический ДНК-связывающий домен с неспецифической нуклеазой. проц. Натл акад. наук США. 2005;102:15797–15802. [Бесплатная статья PMC] [PubMed] [Google Scholar]
9. Xu Y, Lunnen KD, Kong H. Разработка эндонуклеазы N.AlwI, вызывающей разрыв, путем замены домена. проц. Натл акад. наук США. 2001;98:12990–12995. [Бесплатная статья PMC] [PubMed] [Google Scholar]
10. Bitinaite J, Wah DA, Aggarwal AK, Schildkraut I. Для расщепления ДНК требуется димеризация FokI. проц. Натл акад. наук США. 1998;95:10570–10575. [Бесплатная статья PMC] [PubMed] [Google Scholar]
11. Сандерс К.Л., Катто Л.Е., Беллами С.Р., Хэлфорд С.Е. Нацеливание отдельных субъединиц эндонуклеазы рестрикции FokI на определенные цепи ДНК. Нуклеиновые Кислоты Res. 2009 г.;37:2105–2115. [Бесплатная статья PMC] [PubMed] [Google Scholar]
12. Xu SY, Zhu Z, Zhang P, Chan SH, Samuelson JC, Xiao J, Ingalls D, Wilson GG. Открытие природных эндонуклеаз Nb.BsrDI и Nb.BtsI и создание вариантов разрыва верхней цепи из BsrDI и BtsI. Нуклеиновые Кислоты Res. 2007; 35: 4608–4618. [PMC free article] [PubMed] [Google Scholar]
Открытие природных эндонуклеаз Nb.BsrDI и Nb.BtsI и создание вариантов разрыва верхней цепи из BsrDI и BtsI. Нуклеиновые Кислоты Res. 2007; 35: 4608–4618. [PMC free article] [PubMed] [Google Scholar]
13. Armalyte E, Bujnicki JM, Giedriene J, Gasiunas G, Kosinski J, Lubys A. Mva1269I: эндонуклеаза рестрикции IIS мономерного типа из Micrococcus varians с двумя EcoRI- и FokI-подобные каталитические домены. Дж. Биол. хим. 2005; 280:41584–41594. [PubMed] [Google Scholar]
14. Kuhn H, Франк-Каменецкий MD. Мечение уникальных последовательностей в двухцепочечной ДНК в местах вицинальных разрывов, образованных эндонуклеазами, создающими разрывы. Нуклеиновые Кислоты Res. 2008;36:e40. [Бесплатная статья PMC] [PubMed] [Google Scholar]
15. Kiesling T, Cox K, Davidson EA, Dretchen K, Grater G, Hibbard S, Lasken RS, Leshin J, Skowronski E, Danielsen M. Специфическое для последовательности обнаружение ДНК с использованием амплификации сигнала эндонуклеазы разрыва (NESA) Nucleic Acids Res. 2007;35:e117. [Бесплатная статья PMC] [PubMed] [Google Scholar]
2007;35:e117. [Бесплатная статья PMC] [PubMed] [Google Scholar]
16. Xiao M, Phong A, Ha C, Chan TF, Cai D, Leung L, Wan E, Kistler AL, DeRisi JL, Selvin PR, et al. Быстрое картирование ДНК путем обнаружения флуоресцентной одиночной молекулы. Нуклеиновые Кислоты Res. 2007;35:e16. [Бесплатная статья PMC] [PubMed] [Google Scholar]
17. Chiu J, March PE, Lee R, Tillett D. Сайт-направленный, независимый от лигазы мутагенез (SLIM): методология с одной пробиркой, приближающаяся к 100% эффективности в 4 ч. Нуклеиновые Кислоты Res. 2004;32:e174. [Бесплатная статья PMC] [PubMed] [Google Scholar]
18. Hummel JP, Dreyer WJ. Измерение явления связывания белка с помощью гель-фильтрации. Биохим. Биофиз. Акта. 1962; 63: 530–532. [PubMed] [Google Scholar]
19. Vincze T, Posfai J, Roberts RJ. NEBcutter: программа для расщепления ДНК рестрикционными ферментами. Нуклеиновые Кислоты Res. 2003; 31: 3688–3691. [Статья бесплатно PMC] [PubMed] [Google Scholar]
20. Szomolanyi E, Kiss A, Venetianer P. Клонирование модифицированного гена метилазы Bacillus sphaericus R в Escherichia coli . Ген. 1980;10:219–225. [PubMed] [Google Scholar]
Клонирование модифицированного гена метилазы Bacillus sphaericus R в Escherichia coli . Ген. 1980;10:219–225. [PubMed] [Google Scholar]
21. Zhu Z, Samuelson JC, Zhou J, Dore A, Xu SY. Разработка ферментов, специфичных для разрыва ДНК, из эндонуклеаз рестрикции IIS типа BsaI, BsmBI и BsmAI. Дж. Мол. биол. 2004; 337: 573–583. [PubMed] [Google Scholar]
22. Samuelson JC, Zhu Z, Xu SY. Выделение эндонуклеаз, специфичных для нитей, из рандомизированной библиотеки экспрессии SapI. Нуклеиновые Кислоты Res. 2004; 32:3661–3671. [Бесплатная статья PMC] [PubMed] [Google Scholar]
23. Хейтер Д.Ф., Луннен К.Д., Уилсон Г.Г. Сайт-специфические ДНК-мутанты гетеродимерной рестрикционной эндонуклеазы R.BbvCI. Дж. Мол. биол. 2005; 348: 631–640. [PubMed] [Google Scholar]
24. Higgins LS, Besnier C, Kong H. Эндонуклеаза никинга N.BstNBI тесно связана с эндонуклеазами рестрикции II типа MlyI и PleI. Нуклеиновые Кислоты Res. 2001; 29: 2492–2501. [PMC free article] [PubMed] [Google Scholar]
25.